关键词 喉鳞状细胞癌;非编码RNA;ceRNA
Abstract Objective: To construct a ceRNA regulatory network of laryngeal squamous cell carcinoma using GEO database, and to explore the key miRNAs of laryngeal squamous cell carcinoma. Methods: The high-throughput sequencing data of GSE137308 in the GEO database were used to analyze the abnormal expression of miRNA, lncRNA and mRNA in laryngeal squamous cell carcinoma (|logFC|>1, p<0.05). The lncRNA-miRNA-mRNA regulatory network of laryngeal squamous cell carcinoma was constructed according to the ceRNA regulatory network theory and visualized using Cytoscape software. The key miRNAs of laryngeal squamous cell carcinoma were delineated based on the number of nodes in the ceRNA regulatory network. Results: In laryngeal squamous cell carcinoma, 1342 differentially expressed mRNAs were found, of which 926 were up-regulated and 416 were down-regulated; 25 differentially expressed miRNAs were up-regulated by 15 and down-regulated by 10; 989 differentially expressed lncRNAs, of which up-regulated 644, down 345. The ceRNA regulatory network of laryngeal squamous cell carcinoma was constructed and two key miRNAs of laryngeal squamous cell carcinoma (hsa-miR-1299, hsa-miR-6720-5p) were found.
Key words laryngeal squamous cell carcinoma; non-coding RNA; ceRNA
头颈部鳞癌是全球第六大常见恶性肿瘤,其中喉癌是其最为常见的恶性肿瘤之一,仅次于口腔癌[1]。在喉癌中,喉鳞状细胞癌约占85%~90%[2]。喉鳞状细胞癌的发病诱因有很多,包括不良的生活习惯和人乳头瘤病毒感染等。尽管喉鳞状细胞癌发病早期可以通过手术或者辅助疗法进行治疗,但是其晚期患者的预后并没有得到十分显著的改善。据报道,晚期喉鳞状细胞癌的复发率在25%-50%之间[3]。因此,阐明喉鳞状细胞癌的发病分子机制是十分重要的。
随着测序技术的大面积推广应用,人们对疾病的发生发展认识已经大大提高了。过去的研究主要集中在mRNA上。近几年发现,一些非编码RNA在疾病的发生发展中也起着十分重要的作用[4-5]。Salmena等在2011年提出了竞争内源性RNA(ceRNA)的假说,指出lncRNA、mRNA和其他类型的RNA可以通过竞争性结合miRNA,从而起到调控作用[6]。同时许多研究通过实验证明了这一观点,例如lncRNA TUG1可以竞争性地与miR-335-5p结合以调节ROCK1,随后诱导骨肉瘤细胞的迁移和侵袭[7]。 ceRNA的研究已有很多,但是喉鳞状细胞癌的ceRNA调控网络机制仍未完全阐明。本文利用GEO在线数据库分析喉鳞状细胞癌组织差异表达的lncRNA、mRNA、miRNA,构建ceRNA调控网络,为研究喉鳞状细胞癌的发病分子机制奠定基础。
1 资料与方法
1.1 数据下载与差异表达分析
GSE137308是喉鳞状细胞癌的lncRNA、mRNA、miRNA测序数据GEO编号,共有12组数据,miRNA有6例,包括三例正常组织和三例喉鳞状细胞癌组织;mRNA和lncRNA共用6例,包括三例正常组织和三例喉鳞状细胞癌组织。在NCBI GEO(https://www.ncbi.nlm.nih.gov/geo)数据库中检索GSE137308,用Aspera下载测序数据,然后使用HISAT2、SAMtools、HTSeq、DESeq2、miRDeep2等生物信息学分析软件处理测序数据,差异表达的mRNA、miRNA、lncRNA筛选标准为:|logFC|>1,且P<0.05。
1.2 ceRNA调控网络的构建与关键miRNAs挖掘
使用miRwalk 3.0(http://mirwalk.umm.uni-heidelberg.de/)在线软件预测差异表达的miRNA作用的靶基因,然后取靶基因与差异表达的mRNA的交集作为lncRNA-miRNA-mRNA(ceRNA)网络中的mRNA,miRwalk 3.0的预测标准为:miRDB 与TargetScan能够同时预测到靶基因。使用LncBase Predicted v.2(http://carolina.imis.athena-innovation.gr/)在线软件预测差异表达的miRNA作用的靶lncRNA,然后取靶lncRNA与差异表达的lncRNA的交集作为lncRNA-miRNA-mRNA(ceRNA)网络中的lncRNA,LncBase Predicted v.2的预测标准为:Threshold=0.7。
Cytoscape是一个可视化分子相互作用网络和生物途径的软件平台, 常用于构建lncRNA-miRNA-mRNA、蛋白相互作用、miRNA-mRNA等生物学网络。在lncRNA-miRNA-mRNA网络中,miRNA的节点度可以作为评价miRNA重要性的关键因素。我们使用Cytoscape软件构建lncRNA-miRNA-mRNA网络,根据miRNA的节点度筛选关键的miRNAs。
2 结果
2.1 数据差异分析
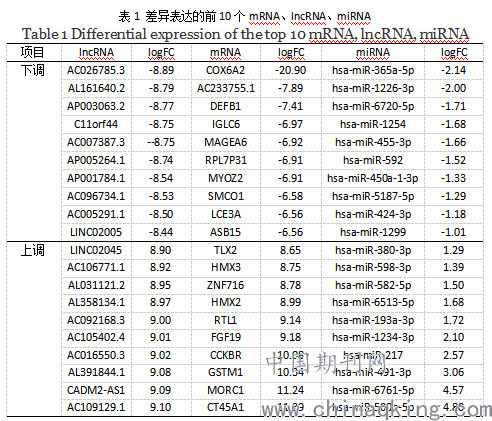
按照|logFC|>1且p<0.05的标准,筛选喉鳞状细胞癌与正常组织的差异表达lncRNA、mRNA、miRNA。结果显示得到差异表达的mRNA 有1342个,其中上调926个,下调416个;差异表达的miRNA有 25个,其中上调15个,下调10个;差异表达的有lncRNA 989个,其中上调644个,下调345个。差异表达的前10个mRNA、lncRNA、miRNA见表1(上下调各10个)。
2.2 ceRNA调控网络的构建与关键miRNA的挖掘
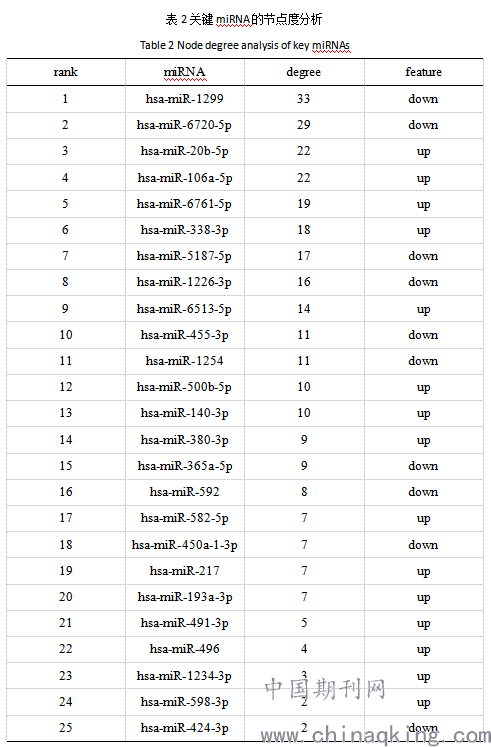
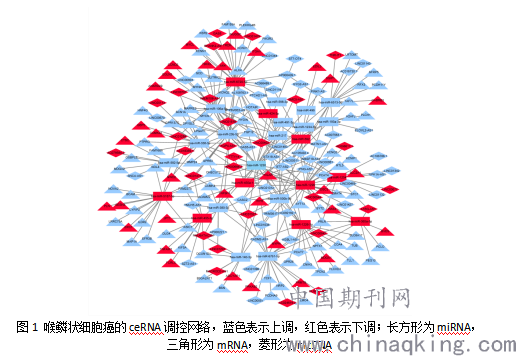
使用Cytoscape软件构建lncRNA-miRNA-mRNA的ceRNA调控网络(图1),同时计算出了lncRNA-miRNA-mRNA网络中miRNA的节点度(表2)。我们发现两个miRNAs(hsa-miR-1299、hsa-miR-6720-5p)具有非常高的节点度,因而认为它们可能与喉鳞状细胞癌的发生发展高度相关。
.png)
3 讨论
喉癌是头颈部最常见的恶性肿瘤之一,其中大多数为喉鳞状细胞癌。近年来,随着医疗技术的不断提高,早期喉癌的治疗效果尚可,但晚期喉癌患者的预后并不理想,5年总生存率<50%,而转移是导致晚期喉癌患者预后不良的主要因素[8]。因此,详细了解喉癌转移的分子机制是开发新型靶向药物的关键。
最近,越来越多的非编码RNA(包括miRNA,lncRNA)被证明在许多疾病(尤其是癌症)的发生发展过程中起到至关重要的调节作用[9-10]。miRNA作为研究最多的非编码RNA,已经显示可以调节人类60%的基因[11]。已经发现许多表达异常的miRNA在癌症的发生发展过程中调节癌基因或抑癌基因。最新的研究表明lncRNA在肿瘤的发生中可以作为一种新型的生物标志物[12]。尽管其分子机制尚未完全了解,但是根据ceRNA理论,lncRNA与mRNA共同竞争结合miRNA,从而在转录后水平调控靶基因。ceRNA调控网络的分析可以扩展我们对编码和非编码RNA功能的认识。为了构建喉鳞状细胞癌的ceRNA调控网络,我们首次使用了GEO数据库中的二代测序数据,同时探讨了3对喉鳞状细胞癌和相邻正常组织中的lncRNA、miRNA、mRNA的差异表达情况。通过生物信息学分析,我们发现了1342个差异表达mRNA,其中上调926个,下调416个;25个差异表达的miRNA,其中上调15个,下调10个;989个差异表达的lncRNA,其中上调644个,下调345个。最后我们根据lncRNA-miRNA-mRNA网络中miRNA的节点数,发现了两个miRNAs(hsa-miR-1299、hsa-miR-6720-5p)具有非常高的节点度,因而认为它们可能与喉鳞状细胞癌的发生发展高度相关。hsa-miR-1299在多种癌症的发生发展中起着至关重要的作用,在乳腺癌[13]、成胶质细胞癌[14]、卵巢癌[15]等癌症中均检测到其异常表达。hsa-miR-6720-5p的相关研究较少,已有研究表明CLSTN3是hsa-miR-6720-5p的作用靶基因[16],而CLSTN3又与癌症的发生发展相关[17-18],故而可以推断hsa-miR-6720-5p与癌症的发生发展也存在一定的关联。下一步我们会收集临床样本,进行分子实验验证hsa-miR-1299和hsa-miR-6720-5p的真实性,进一步的深入研究喉鳞状细胞癌的发病分子机制。
[1] 于华,辛玉芬,段晓东,朱立新,金德钧.喉癌的流行病学病因学动态分析[J].现代生物医学进展,2007(03):393-395.
[2] Genden E M, Ferlito A, Silver C E, et al. Evolution of the management of laryngeal cancer[J]. Oral oncology, 2007, 43(5): 431-439.
[3] Fan L, Zhang A, Deng P. LMX1B mRNA expression and its gene body CpG methylation are valuable prognostic biomarkers for laryngeal squamous cell carcinoma[J]. Biomedicine & Pharmacotherapy, 2019, 117: 109174.
[4] Sanchez Calle A, Kawamura Y, Yamamoto Y, et al. Emerging roles of long non‐coding RNA in cancer[J]. Cancer science, 2018, 109(7): 2093-2100.
[5] Takahashi R, Prieto‐Vila M, Kohama I, et al. Development of miRNA‐based therapeutic approaches for cancer patients[J]. Cancer science, 2019, 110(4): 1140.
[6] Salmena L, Poliseno L, Tay Y, et al. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?[J]. Cell, 2011, 146(3): 353-358.
[7] Wang Y, Yang T, Zhang Z, et al. Long non‐coding RNA TUG 1 promotes migration and invasion by acting as a ce RNA of miR‐335‐5p in osteosarcoma cells[J]. Cancer science, 2017, 108(5): 859-867.
[8] Peyvandi H, Peyvandi A A, Safaei A, et al. Introducing potential key proteins and pathways in human laryngeal cancer: a system biology approach[J]. Iranian journal of pharmaceutical research: IJPR, 2018, 17(1): 415.
[9] Chew C L, Conos S A, Unal B, et al. Noncoding RNAs: master regulators of inflammatory signaling[J]. Trends in molecular medicine, 2018, 24(1): 66-84.
[10] Cosetti M, Yu G P, Schantz S P. Five-year survival rates and time trends of laryngeal cancer in the US population[J]. Archives of otolaryngology–head & neck surgery, 2008, 134(4): 370-379.
[11] Friedman R C, Farh K K H, Burge C B, et al. Most mammalian mRNAs are conserved targets of microRNAs[J]. Genome research, 2009, 19(1): 92-105.
[12] Sahu A, Singhal U, Chinnaiyan A M. Long noncoding RNAs in cancer: from function to translation[J]. Trends in cancer, 2015, 1(2): 93-109.
[13] Chengchen X U, Zhu G, Wang X, et al. Expression profile of microRNAs under different lever of HER-2 in human breast cancer MCF-7 cells[J]. Chinese Journal of Clinical and Experimental Pathology, 2014 (9): 967-970,974.
[14] Dong L, Li Y, Han C, et al. miRNA microarray reveals specific expression in the peripheral blood of glioblastoma patients[J]. International journal of oncology, 2014, 45(2): 746-756.
[15] Kumar S, Kumar A, Shah P P, et al. MicroRNA signature of cis-platin resistant vs. cis-platin sensitive ovarian cancer cell lines[J]. Journal of ovarian research, 2011, 4(1): 17.
[16] Ren H, Liu Z, Liu S, et al. Profile and clinical implication of circular RNAs in human papillary thyroid carcinoma[J]. PeerJ, 2018, 6: e5363.
[17] Johanneson B, Chen D, Enroth S, et al. Systematic validation of hypothesis-driven candidate genes for cervical cancer in a genome-wide association study[J]. Carcinogenesis, 2014, 35(9): 2084-2088.
[18] J?nsson G, Staaf J, Vallon-Christersson J, et al. Genomic subtypes of breast cancer identified by array-comparative genomic hybridization display distinct molecular and clinical characteristics[J]. Breast Cancer Research, 2010, 12(3): R42.
论文作者:徐后喜
论文发表刊物:《世界复合医学》2019年9期
论文发表时间:2020/1/16
标签:细胞论文; 差异论文; 网络论文; 在线论文; 序数论文; 发生论文; 节点论文; 《世界复合医学》2019年9期论文;
